







Методов выделения микробной ДНК на сегодня существует очень много, однако они не приспособлены под работу с морскими образцами. Отсутствие понятной, отработанной методики для этой задачи замедляет исследование морских микроорганизмов — при этом именно с таких исследований может начаться поиск новых генов, отвечающих за синтез антибиотиков или ферментов генетических редакторов. Коллектив ученых из Сколтеха, МГУ имени М. В. Ломоносова и других ведущих научных организаций во главе с руководителем Лаборатории анализа метагеномов Артемом Исаевым выявил наиболее эффективные наборы для выделения ДНК из разных типов морских образцов и представил результаты в новой работе в журнале Scientific Reports.
Акватории северных морей России долгое время оставались неисследованными. «Это труднодоступная зона, но очень разнообразная, поэтому мы сфокусировались именно на Тихом и Северном Ледовитом океанах. Совместно с Центром морских исследований МГУ мы организовали экспедиции и собрали три типа образцов: морские воду и грунт, а также образцы беспозвоночных», — рассказала первый автор работы Алина Демкина, старший лаборант в Лаборатории анализа метагеномов Сколтеха.
По словам ученых, несмотря на то что метагеномные исследования активно развиваются, до сих пор не выработано единых критериев и стандартов по работе с морскими образцами. «У природных образцов есть свои особенности. Нашей целью было выделить из них ДНК, отсеквенировать ее и проверить, какие бактерии там живут. Мы столкнулись с тем, что отработанных методик для выделения ДНК хорошего качества из таких образцов нет. Создавать какой-то протокол с нуля — задача очень долгая, дорогая и трудоемкая, поэтому мы сфокусировались на готовых коммерческих наборах и проверили, какой из них может давать результат наилучшего качества для разных типов морских образцов», — поделилась Дарья Слонова, соавтор работы, младший научный сотрудник в Лаборатории анализа метагеномов Сколтеха.
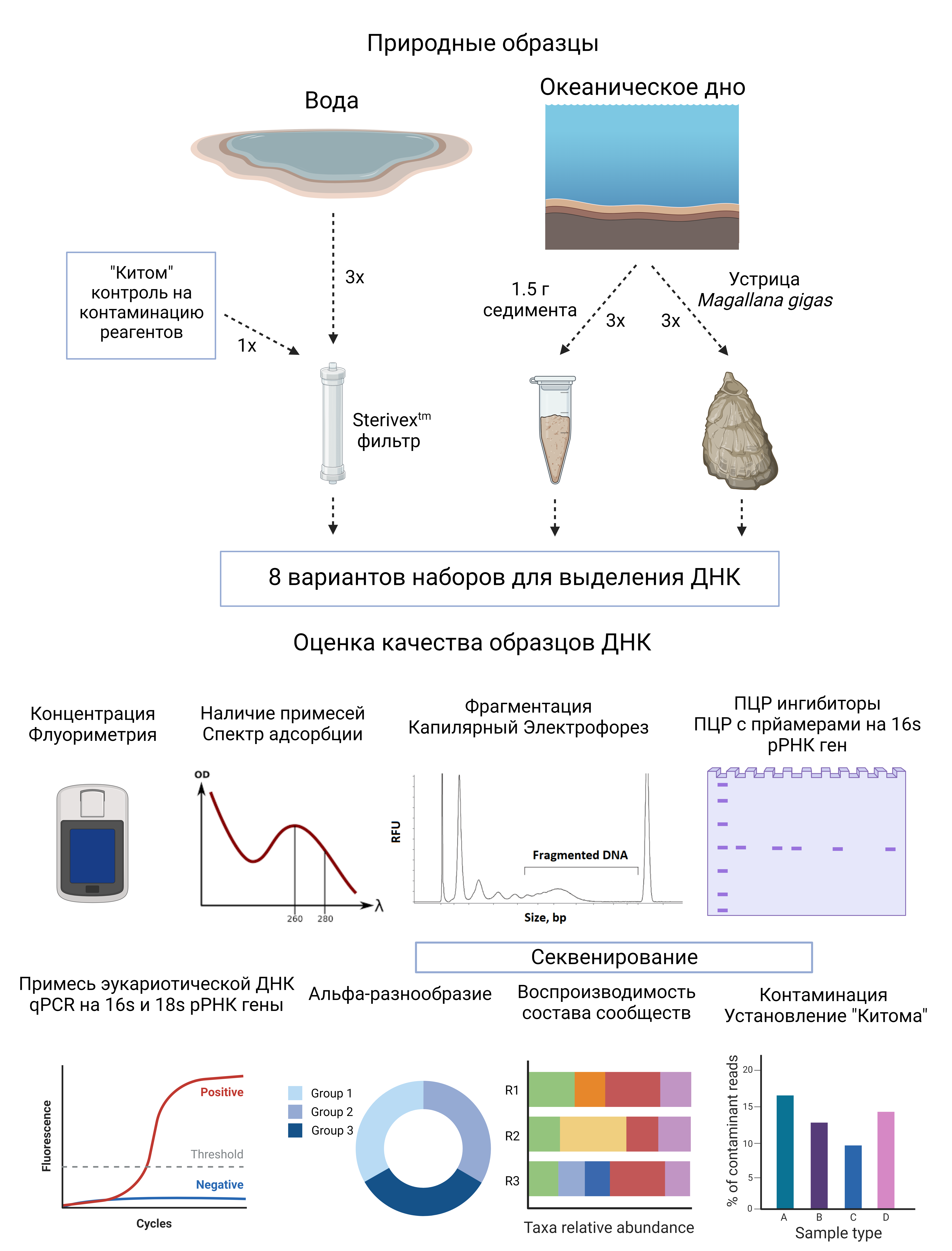
Схема исследования. Источник: Benchmarking DNA isolation methods for marine metagenomics.
Исследователи провели испытания восьми наборов для выделения ДНК. Для каждой комбинации набора и образца измеряли количество очищенной ДНК, степень ее фрагментации, наличие загрязняющих веществ, ингибирующих ПЦР, примесь ДНК эукариот, альфа-разнообразие и воспроизводимость результирующего состава сообщества на основе секвенирования ампликонов 16S рРНК. Также ученые определили так называемые «китомы» (от англ. kit — набор) — состав микробных таксонов, которые могут присутствовать в самих реагентах, используемых для выделения ДНК. Это особенно важно, так как воды северных морей достаточно бедны микробами, и поэтому даже незначительная примесь контаминирующей ДНК может отразиться на анализе.
«В статье мы даем рекомендации ученым, какие методы лучше всего подходят, чтобы правильно выделить ДНК из морских образцов», — продолжила Алина Демкина. «Исследователи смогут увидеть в статье, какие наборы подходят под каждый конкретный тип образца, в зависимости от целей их работы», — поддержала Дарья Слонова.

